

HelixFold类别文心·生物计算大模型应用蛋白结构预测模型概述HelixFold端到端地学习蛋白质结构,通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。利用大规模无标注数据强化蛋白质表示能力,构建的全国产化软硬件适配的高效蛋白质结构分析大模型,在国产DCU环境下训练千万级别蛋白仅需到2.6
免费申请测试 >> *价格优惠政策请联系客服咨询HelixFold端到端地学习蛋白质结构,通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。
预测蛋白结构时,会利用氨基酸序列信息在蛋白质库中搜索多序列比对(MSA)。MSA 可以反映氨基酸序列中的保守性区域(即不容易产生突变),这些保守性区域和蛋白质的结构息息相关,比如可能被折叠在蛋白质内层,不容易和外界产生相互作用,进而不易受影响发生突变。
在飞桨强大的高性能并行计算能力支持下,飞桨螺旋桨 PaddleHelix 生物计算团队发布了蛋白结构预测模型 HelixFold,围绕着显存峰值、训练速度、分布式策略进行了全面性能优化。
HelixFold
类别文心·生物计算大模型
应用蛋白结构预测
模型概述
HelixFold端到端地学习蛋白质结构,通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。利用大规模无标注数据强化蛋白质表示能力,构建的全国产化软硬件适配的高效蛋白质结构分析大模型,在国产DCU环境下训练千万级别蛋白仅需到2.6天,在 GPU 上相同硬件环境配置下,训练性能和部分场景效果显著优于 AlphaFold2。
模型说明
蛋白质是一切生命活动的基础,它几乎参与了所有的生物学过程。如遗传、发育、繁殖等等。对蛋白质进行深入地研究,能让我们从更深层次诠释生命体的构成和运作变化规律,进而全面揭示生命运行、发展的机制,激发生物科学、药物研发、合成生物学、酶科学等领域的发展。因此,探究生物体内各种蛋白质的功能及其机制等是目前蛋白质研究的主要内容,同时也是后基因组时代生命科学领域的主要研究热点之一。蛋白质的功能很大程度上取决于蛋白质的结构,因此如何破解蛋白质的三维结构成为了科学家研究的重点。
在 2020 年的 CASP 14 上,谷歌 DeepMind 团队的 AlphaFold2 以惊人的 92.4 分登顶第一, 2021 年 7 月 15 日, DeepMind 团队在《Nature》上发表论文详细描述了 AlphaFold2 的设计思路,并提供了可供运行的基于 JAX 的模型和代码。考虑到 JAX 受众偏向专业的 AI 科学计算研究人员,且飞桨社区尚没有蛋白质结构预测相关的开源项目,百度螺旋桨 PaddleHelix 生物计算团队,基于飞桨深度学习框架 paddlepaddle ,在 AlphaFold2 模型基础上,针对孤蛋白进行优化,同时对模型性能进行优化,开发出可以在纯国产软硬件上训练的蛋白结构预测模型 HelixFold ,提供给广大飞桨开发者使用,帮助大家快速入门蛋白质结构预测。
HelixFold 端到端地学习蛋白质结构,通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。利用大规模无标注数据强化蛋白质表示能力,构建的全国产化软硬件适配的高效蛋白质结构分析大模型,在国产 DCU 环境下训练千万级别蛋白仅需到 2.6 天,在 GPU 上相同硬件环境配置下,训练性能和部分场景效果显著优于 AlphaFold2。
HelixFold 创新性的提出分支并行(Branch Parallelism, BP)策略,将不同的网络模型分支放在不同的卡上并行计算,从而在 initial training 阶段大幅提高了模型并行效率和训练速度。并且,分支并行与已有的动态轴并行(Dynamic Axial Parallelism, DAP)和数据并行(Data Parallelism,DP)结合使用,通过 BP-DAP-DP 三维混合并行,进一步加快了模型的整体训练速度。
针对 AlphaFold2 中 Gated Self-Attention 小算子组合 CPU 调度开销大、模型参数小、参数个数多的问题,HelixFold 将 Gated Self-Attention 整个模块融合用一个算子实现,将 CPU 调度开销优化到极致。同时,将数千个小张量融合成一个连续的大张量,模型参数的梯度、优化器状态都相应更新,大幅减少了访存次数、CPU 调度开销和显存碎片,从而提升了训练速度。
采用 Recompute、BFloat16、显存复用、Subbatch(Chunking)等技术,将显存峰值降低到 40G 以内,同时支持 MSA 长度为 512、ExtraMSA 长度为 5120、残基序列长度为 384 的最大模型配置的微调训练,从而解决了模型结构深,中间结果计算量大,ExtraMSAStack 输入过长等导致无法训练的问题。
预测蛋白结构时,会利用氨基酸序列信息在蛋白质库中搜索多序列比对(MSA)。MSA 可以反映氨基酸序列中的保守性区域(即不容易产生突变),这些保守性区域和蛋白质的结构息息相关,比如可能被折叠在蛋白质内层,不容易和外界产生相互作用,进而不易受影响发生突变。
借鉴 AlphaFold2 的多轨机制:1. 对 MSA 进行建模,根据多条蛋白序列间的联系推理共进化信息;2. 对氨基酸对间的关系建模学习几何空间的三角约束信息,推理出空间信息。多轨机制得到的蛋白质表征接入到结构学习模块,端到端地解码出蛋白质中每个重原子(C,N,O,S)的坐标。
#加载环境 wget https://baidu-nlp.bj.bcebos.com/PaddleHelix/HelixFold/paddlepaddle_gpu-0.0.0-cp37-cp37m-linux_x86_64.whl sh setup_env conda activate helixfold # activate the conda environment #下载数据集和模型参数 scripts/download_all_data.sh <DOWNLOAD_DIR> #进行预测 fasta_file="target.fasta" # path to the target protein model_name="model_1" # the alphafold model name DATA_DIR="data" # path to the databases OUTPUT_DIR="helixfold_output" # path to save the outputs
python3 run_helixfold.py
–fasta_paths=${fasta_file}
–data_dir=${DATA_DIR}
–small_bfd_database_path=${DATA_DIR}/small_bfd/bfd-first_non_consensus_sequences.fasta
–uniref90_database_path=${DATA_DIR}/uniref90/uniref90.fasta
–mgnify_database_path=${DATA_DIR}/mgnify/mgy_clusters_2018_12.fa
–pdb70_database_path=${DATA_DIR}/pdb70/pdb70
–template_mmcif_dir=${DATA_DIR}/pdb_mmcif/mmcif_files
–obsolete_pdbs_path=${DATA_DIR}/pdb_mmcif/obsolete.dat
–max_template_date=2020-05-14
–model_names=${model_name}
–output_dir=${OUTPUT_DIR}
–preset=‘reduced_dbs’
–jackhmmer_binary_path /opt/conda/envs/helixfold/bin/jackhmmer
–hhblits_binary_path /opt/conda/envs/helixfold/bin/hhblits
–hhsearch_binary_path /opt/conda/envs/helixfold/bin/hhsearch
–kalign_binary_path /opt/conda/envs/helixfold/bin/kalign
–random_seed=0
https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/protein_folding/helixfold
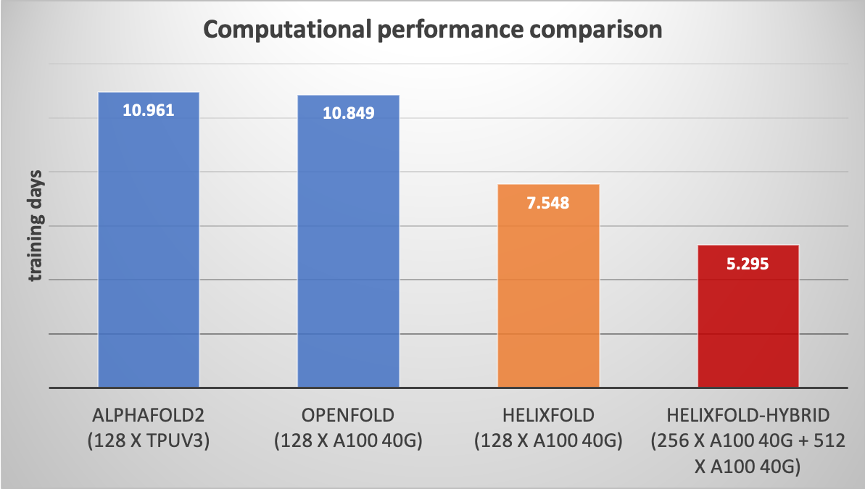
在飞桨强大的高性能并行计算能力支持下,飞桨螺旋桨 PaddleHelix 生物计算团队发布了蛋白结构预测模型 HelixFold,围绕着显存峰值、训练速度、分布式策略进行了全面性能优化。通过与原版 AlphaFold2 模型和哥伦比亚大学 Mohammed AlQuraishi 教授团队基于 PyTorch 复现的 OpenFold 模型的性能对比测试显示,HelixFold 模型的训练性能相比 AlphaFold2 提升 106.97%,相比 OpenFold 提升 104.86%。

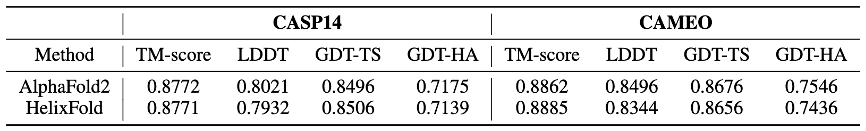
在性能大幅度提升的同时,HelixFold 从头端到端完整训练可以达到 AlphaFold2 论文媲美的精度。在包含 87 个蛋白的 CASP14 数据集和包含 371 个蛋白的 CAMEO 数据集上,HelixFold 模型 TM-score 指标分别达到 0.8771 和 0.8885,与原版 AlphaFold2 准确率相当甚至更优

微信公众号

手机站
COPYRIGHT 2007-2020 TUIDC ALL RIGHTS RESERVED 腾佑科技-百度AI人工智能_百度人脸识别_图像识别_语音识别提供商
地址:河南省郑州市姚砦路133号金成时代广场6号楼13层 I CP备案号:豫B2-20110005-1 公安备案号: 41010502003271
声明:本站发布的内容版权归郑州腾佑科技有限公司所有,本站部分素材来源于网络及网友投稿,若无意中侵犯了您的版权,请致电在线客服我们将在核实后予以删除!